I. Introduction : le génome et l’épigénome1
Les caractéristiques d’un être vivant sont contrôlées à la fois par son environnement et par son information génétique. L’information génétique est l’ensemble des instructions mises en œuvre dans la fabrication, ou la croissance, d’un être vivant : elle permet, par exemple, à un animal de fabriquer davantage de lui-même à partir d’une nourriture qui est de constitution différente de la sienne (le lapin fabrique de la matière « lapin » en mangeant des carottes). C’est donc une information qui se trouve dans l’organisme vivant lui-même, et qui le définit suffisamment pour qu’il puisse croître, se réparer, ou se reproduire, pratiquement à l’identique, malgré la variabilité du milieu extérieur.
Matériellement, cette information est portée par les molécules d’ADN (acide désoxyribonucléique), qui se trouvent dans pratiquement toutes les cellules2. Les molécules d’ADN portent une séquence de groupements chimiques (cytosine, guanine, adénine et thymine, habituellement symbolisés par leurs initiales : C, G, A et T ; voir Figure 1) : c’est cette séquence, qui constitue l’information génétique (une séquence « ACCGAT » n’est pas la même que « GACTTA », ces deux séquences portent donc une information différente).
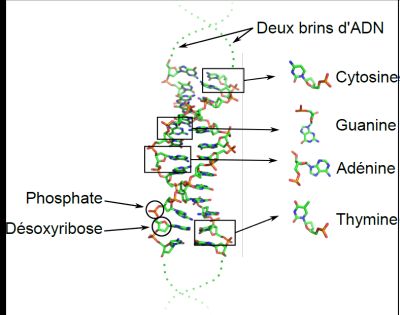
Fig. 1 : La structure de l’ADN.
La molécule d’ADN forme une longue double hélice, dont les deux brins s’enroulent l’un autour de l’autre (ici, juste un extrait de la structure est représenté : les deux brins s’étendent de part et d’autre, selon la direction des pointillés). Chaque atome est représenté par une couleur (phosphore en orange, carbone en vert, oxygène en rouge, azote en bleu ; les atomes d’hydrogène ne sont pas représentés). L’extérieur de la double hélice est constitué d’une répétition monotone des mêmes groupements d’atomes (formant des phosphates et des désoxyriboses, en alternance), mais l’intérieur est une succession de groupements d’atomes variables (la cytosine, la guanine, l’adénine et la thymine, détaillés sur la droite).
Des machineries cellulaires spécialisées copient la séquence de l’ADN en une molécule de nature chimique très proche, l’ARN (acide ribonucléique). Certains ARN ont une activité biologique par eux‑mêmes ; d’autres servent de programmes dirigeant la synthèse des protéines (la molécule d’ARN est lue par une machinerie cellulaire qui fabrique une protéine dont la nature dépend de la séquence de l’ARN). In fine, c’est l’activité moléculaire des ARN et des protéines qui contrôle les caractéristiques de la cellule, en lui donnant ses propriétés mécaniques, en gouvernant sa composition chimique, en lui conférant une sensibilité à certains signaux extra‑cellulaires...
On appelle « génome » la totalité de la séquence d’ADN d’un individu (à de très rares exceptions près, toutes les cellules d’un même individu portent la même séquence d’ADN). Chez les animaux et les plantes, seule une petite partie du génome semble indiscutablement fonctionnelle (de nombreuses régions génomiques sont même rarement copiées sous forme d’ARN, il n’est pas clair qu’elles jouent un rôle biologique). Les régions fonctionnelles sont dispersées le long du génome, en de petites unités individuelles appelées « gènes ». Les gènes sont donc des modules fonctionnels : un gène contient l’information qui permet la synthèse d’un ARN ou d’une protéine (parfois : de plusieurs ARN ou protéines similaires entre eux), et cet ARN ou protéine conférera à la cellule une propriété particulière. C’est ainsi que, par exemple, certains gènes humains sont connus pour déterminer la couleur des yeux, le groupe sanguin, la tolérance à certains aliments...
Les gènes sont exprimés ou non, selon les types cellulaires (les gènes des kératines sont fortement exprimés dans les cellules de la peau : ces cellules vont donc se remplir de kératines, protéines qui leur confèrent leur résistance mécanique ; les neurones expriment les gènes de protéines réceptrices qui les rendent sensibles aux signaux émis par d’autres neurones ; etc.). Il existe une grande variété de mécanismes de contrôle de l’expression des gènes, qui allument ou éteignent leur expression selon le type cellulaire, et selon les stimuli reçus de l’extérieur de la cellule.
Parmi ces mécanismes, certains contrôlent la copie de l’ADN en ARN, d’autres contrôlent la synthèse de protéine à partir d’un ARN, d’autres contrôlent l’activité des protéines elles‑mêmes. La plupart de ces mécanismes impliquent la reconnaissance d’un gène‑cible ou d’un ARN‑cible par une protéine régulatrice (qui, en interagissant avec ses molécules‑cibles, va moduler leur expression). Notamment, il est établi que des modifications chimiques de l’ADN, ou des protéines qui lui sont associées, a tendance à affecter l’expression des gènes du voisinage. On appelle « épigénome » l’ensemble des modifications chimiques portées par l’ADN ou par ses protéines associées. L’épigénome est déposé par des protéines spécifiques, qui reconnaissent des régions‑cibles sur le génome, et affectent leur expression en déposant des modifications chimiques particulières sur l’ADN ou les protéines qui lui sont associées.
II. L’édition du génome
Une grande partie du travail des généticiens consiste à identifier les fonctions biologiques des gènes. Alors qu’il est facile de mesurer l’expression des gènes (donc, de déterminer dans quelles cellules ils sont copiés sous forme d’ARN, et de comparer quantitativement les niveaux d’expression dans des types cellulaires différents), il est souvent plus difficile de comprendre le mode d’action des ARN ou protéines produits par les gènes. Une méthode d’analyse convaincante consiste à inactiver ce gène (par exemple, en modifiant sa séquence pour le rendre incapable de guider la production d’une protéine, ou pour le rendre impropre à la copie sous forme d’ARN), pour ensuite évaluer les conséquences de cette inactivation sur la biologie de l’organisme. Modifier un fragment du génome, en remplaçant sa séquence par une séquence choisie par l’expérimentateur, est appelé « éditer le génome ». De la même manière qu’un éditeur de texte permet, sur ordinateur, de modifier un document de texte, « éditer » le génome consiste à modifier le « texte », constitué d’une succession de A, C, G et T, dans le génome.
Jusqu’à récemment, l’édition du génome était très difficile chez la plupart des organismes. Alors que des méthodes simples et efficaces existaient depuis longtemps pour modifier le génome de la levure (un champignon unicellulaire), la modification contrôlée du génome était d’une grande difficulté chez les animaux et les plantes. Difficile, mais pas tout à fait impossible : il était possible d’utiliser un mécanisme naturel de réparation de l’ADN, appelé « recombinaison homologue ». Ce phénomène est très répandu chez les espèces vivantes : il permet aux cellules de réparer l’ADN lorsqu’il a subi un dommage, en prenant comme modèle une autre molécule d’ADN de séquence similaire (sur la molécule endommagée, le dommage est remplacé par un fragment d’ADN copié sur l’ADN‑modèle). Pour éditer une séquence donnée sur le génome, il faut donc commencer par couper l’ADN‑cible, tout en fournissant à la cellule un ADN de séquence similaire, mais contenant la séquence qu’on souhaite voir introduite. La machinerie endogène de la cellule réparera la coupure et, avec une probabilité qui dépend de la quantité d’ADN‑modèle introduit, elle utilisera parfois l’ADN‑modèle fourni par l’expérimentateur.
Il faut signaler que ce mode de réparation de l’ADN n’est pas le seul : outre la réparation par recombinaison homologue, les cellules peuvent réparer leur ADN par un autre processus, où la coupure est réparée par un simple raboutage des deux fragments, sans intervention d’un ADN‑modèle (voir Figure 2).
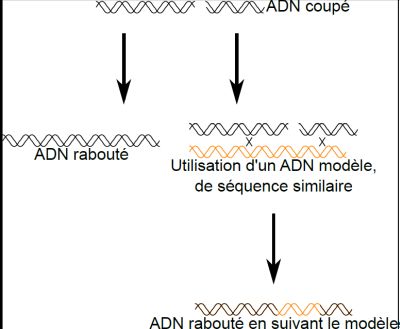
Fig. 2 : Les processus naturels de réparation de coupure de l’ADN peuvent être mises à profit pour l’édition du génome.
Lorsque, suite à un accident quelconque, l’ADN est coupé, des machineries cellulaires spécialisées peuvent réparer la coupure selon plusieurs mécanismes. À gauche : dans le premier mécanisme, appelé non-homologous end joining, les deux fragments sont simplement raboutés l’un à l’autre (si ce processus est lent, il arrive que les fragments soient partiellement modifiés par des enzymes cellulaires avant le raboutage, ce qui peut conduire à l’apparition de petites délétions ou insertions de séquence à l’endroit de la coupure). À droite : dans le deuxième mécanisme, appelé « recombinaison homologue », la machinerie de réparation utilise un ADN-modèle (ici en orange), dont la séquence contient de longs segments d’identité à celle de l’ADN à réparer, et elle copie cet ADN‑modèle dans l’intervalle à rabouter. Si cet ADN‑modèle a été fourni par l’expérimentateur, ce processus de réparation peut aboutir à l’introduction d’une séquence choisie arbitrairement, à l’endroit de la coupure.
La recombinaison homologue offre donc la possibilité d’introduire une séquence quelconque, choisie par l’expérimentateur, à l’endroit où l’ADN a été coupé. Mais il faut avoir réussi à couper l’ADN à l’endroit que l’on désire muter, sans modifier le reste du génome : c’était la plus grande difficulté de l’exercice.
Jusqu’à récemment, il existait deux solutions pour couper le génome à un endroit spécifique, mais ces méthodes présentaient de sérieuses limitations :
- On connaît quelques enzymes (les « méganucléases ») qui coupent l’ADN quand il porte une séquence bien spécifique, et assez longue (de l’ordre d’une vingtaine de caractères A, C, G et T). Le motif de séquence reconnu par ces enzymes est tellement long qu’il est très rare dans les génomes (en général, ces enzymes n’ont pas plus d’un site de reconnaissance par génome), ce qui garantit qu’elles ne couperont l’ADN qu’à un seul endroit... mais ce qui implique, également, que l’expérimentateur n’aura pas le choix de l’endroit où l’enzyme coupera. S’il veut muter un gène donné, il y a très peu de chances que l’unique site de coupure génomique de l’enzyme se trouve dans son gène d’intérêt ;
- On commence à connaître assez bien les règles de reconnaissance de l’ADN par les protéines. Il est donc possible, dans une certaine mesure, de produire des enzymes artificielles, protéines dont la nature aura été choisie pour qu’elles se fixent sur une séquence d’ADN d’intérêt, et la coupent. La production de ces enzymes est longue et difficile, et puisque les prédictions de leur interaction avec l’ADN ne sont pas parfaites, il n’est pas rare qu’elles n’aient pas la spécificité souhaitée, pour la séquence d’intérêt.
Finalement, tant que d’autres outils n’étaient pas disponibles, l’édition du génome restait peu utilisée.
III. CRISPR/Cas : un système immunitaire bactérien
La découverte du système CRISPR/Cas9 découle de l’étude d’un système de défense des bactéries contre leurs pathogènes. Pour nous, humains, les bactéries sont parfois des pathogènes, mais il faut savoir qu’elles‑mêmes, sont soumises aux agressions de pathogènes qui leur sont spécifiques (principalement, des virus spécifiques des bactéries – on les appelle des « bacté‑rio‑phages » – et des ADN invasifs, sortes de parasites moléculaires, qui se reproduisent dans la cellule bactérienne à son détriment).
Certaines bactéries (à peu près la moitié des espèces étudiées) possèdent, à un endroit de leur génome, des séquences répétées à intervalles réguliers. Chacune des copies se trouve être, de plus, une séquence « palindromique » (c’est‑à‑dire qu’elle est identique si on la lit de gauche à droite, ou de droite à gauche). Ces curieuses répétitions génomiques, identifiées dans les années 1980 et 1990, ont donc été baptisées « CRISPR », pour « Clustered Regularly Interspaced Short Palindromic Repeats » (soit : répétitions palindromiques courtes, regroupées et régulièrement réparties). Entre les copies répétées se trouvaient des séquences qui, elles, étaient différentes entre elles (voir Figure 3).
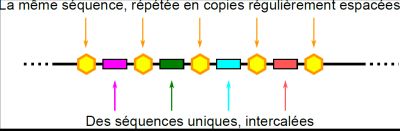
Fig. 3 : Structure des répétitions CRISPR dans un génome bactérien.
Les hexagones jaunes symbolisent les copies de la séquence palindromique et les rectangles colorés représentent les séquences (différentes entre elles) intercalées.
L’observation, en 2005, que ces séquences intercalaires, non‑répétées, provenaient du génome de bactériophages ou d’ADN invasifs, a suggéré qu’elles participaient à un mécanisme de défense contre ces pathogènes. Finalement, c’étaient ces séquences intercalaires (les rectangles colorés sur la Figure 3) qui s’avéraient plus intéressants que les palindromes répétés.
Cette hypothèse a été confirmée en 2007, quand il est apparu qu’on pouvait conférer à une bactérie une résistance face à un pathogène, si on modifiait une séquence intercalaire pour la rendre spécifique de ce pathogène. Cette portion de génome contient donc une sorte de répertoire de séquences de pathogènes, contre lesquels la bactérie sera résistante, et qui constitue une sorte de mémoire immunitaire.
Le mécanisme de cette résistance aux pathogènes a été élucidé dans les années suivantes : des protéines appelées « protéines Cas » (pour : « CRISPR‑associated », associées aux CRISPR) permettent l’introduction de morceaux de séquence de pathogènes dans cette région génomique, quand la cellule est infectée ; d’autres protéines Cas fabriquent des ARN de taille et de repliement spatial homogènes, à partir de ces séquences intercalaires ; ces ARN sont ensuite chargés sur une autre protéine Cas (celle qu’on appelle Cas9) : cet assemblage ARN/protéine est capable de reconnaître les ADN qui portent la même séquence que l’ARN, et de les dégrader. Dans l’assemblage ARN/Cas9, l’ARN joue donc le rôle de guide (il reconnaît les ADN qui partagent sa séquence), et la protéine Cas9 joue le rôle d’effecteur, qui détruit l’ADN‑cible (voir Figure 4).
Fig. 4 : Mode d’action de la protéine Cas9.
L’ARN-guide (ici en rouge) peut reconnaître les ADN qui portent la même séquence que lui : dans l’ADN double-brin, si un brin porte la même séquence que celle de l’ARN, alors l’autre brin d’ADN porte une séquence qui lui est complémentaire. L’ARN est donc capable de s’apparier avec ce deuxième brin, et de former une double hélice ADN/ARN. Cet appariement stabilise l’interaction entre la protéine Cas9 et l’ADN, et laisse le temps à la protéine Cas9 de couper l’ADN à une position spécifique (représentée ici par une étoile bleue). À noter : les protéines Cas9 des différentes bactéries ont également besoin que l’ADN-cible porte un motif de séquence particulier, appelé « PAM » (pour : « protospacer adjacent motif », motif adjacent à la séquence intercalaire). Cette limitation ne permet donc pas au système CRISPR/Cas9 de couper n’importe quel ADN, seulement ceux qui portent un PAM à proximité de la séquence-cible. Mais le PAM est une séquence généralement courte, qui est donc fréquente dans les séquences d’ADN (par exemple, la plupart des expérimentateurs utilisent la protéine Cas9 de la bactérie Streptococcus pyogenes, dont le PAM est la séquence GG : une séquence aussi courte se retrouve fréquemment dans les séquences-cibles potentielles).
La découverte de ce système a permis de mieux comprendre l’immunité des bactéries vis‑à‑vis de leurs pathogènes. Elle a également permis une application biotechnologique utile : puisque Cas9 coupe les ADN qui présentent une séquence identique à celle de l’ARN-guide, il est donc possible de programmer Cas9 pour qu’elle aille couper un ADN-cible quelconque, choisi par l’expérimentateur (il suffit juste de lui fournir un ARN‑guide spécifique de la cible visée). Cas9 est donc une enzyme programmable, qui peut couper une région génomique choisie, et donc débloquer le verrou technologique qui empêchait généralement la mise en œuvre de techniques d’édition du génome (voir section).
IV. Un couteau suisse moléculaire
La découverte du fonctionnement de Cas9 a donc ouvert la voie de l’édition du génome de manière simple : en introduisant, dans les cellules‑cibles, la protéine Cas9 et l’ARN‑guide (ou alors : des ADN portant les gènes de Cas9 et de l’ARN, que la cellule elle-même utilisera pour les fabriquer in situ), ainsi qu’un ADN‑modèle portant la séquence qu’on souhaite voir introduite dans la région génomique visée, il devient possible d’éditer à volonté à peu près n’importe quelle cible génomique. Le choix de la cible génomique reste contraint par la nécessité de la présence d’une séquence PAM dans son voisinage (voir Figure 4), mais les séquences PAM sont si courtes que la contrainte est légère : il est facile de trouver une PAM à proximité de n’importe quelle cible génomique.
Naturellement, il reste encore à pouvoir introduire dans les cellules la protéine Cas9 et l’ARN‑guide, ou un ADN portant leurs gènes, ainsi que l’ADN‑modèle. Mais il se trouve que des protocoles bien optimisés sont disponibles depuis longtemps pour ce genre d’opération : bien avant la découverte du fonctionnement de Cas9, la communauté scientifique avait dû mettre au point des techniques d’introduction d’ADN, d’ARN ou de protéines dans les cellules, pour d’autres types d’expérience. Des protocoles efficaces existaient donc déjà, pour permettre d’introduire tous ces réactifs dans des cellules en culture, ou dans des œufs fécondés. Introduire une modification génétique dans un œuf fécondé (plutôt que dans une cellule adulte) a l’avantage de permettre l’obtention d’un organisme entièrement modifié, sur l’ensemble de ses cellules : puisque l’organisme est issu d’un œuf fécondé, qui s’est divisé de nombreuses fois, chaque cellule de l’organisme final contient le même ADN que l’œuf fécondé initial. Si on a édité le génome de l’œuf fécondé, cette modification du génome se transmettra à toutes les cellules, tout au long du développement, et on obtiendra un adule dont le génome a été modifié sur la totalité de ses cellules.
L’une des limitations de la technique tient à sa spécificité : on aimerait s’assurer que seule la région ciblée, et aucune autre, a été coupée par Cas9. Rien n’est jamais parfait, et il est connu que Cas9 a tendance à reconnaître des séquences d’ADN qui ne sont qu’imparfaitement identiques à celle de son ARN‑guide. Mais des améliorations commencent à être apportées : des versions mutées de la protéine Cas9 (qui ont moins d’affinités pour l’ADN, et qui, donc, dépendent encore plus fortement de la qualité de l’appariement entre l’ARN‑guide et l’ADN‑cible) ont maintenant des spécificités telles qu’il devient difficile de détecter des coupures non‑ciblées, malgré la grande sensibilité des méthodes de mesure.
Rapidement, il est apparu que Cas9 pouvait même servir à d’autres usages que l’édition du génome. On peut la muter, de manière à la rendre incapable de couper l’ADN. Guidée par un ARN, elle va donc reconnaître une région‑cible sur le génome, mais y rester associée sans la couper. Elle peut donc servir de plateforme pour attirer d’autres types d’enzymes sur la région cible. Par exemple, on peut préparer un gène artificiel, qui fusionne le gène Cas9 avec le gène d’une enzyme de modification de l’épigénome (une de ces enzymes qui déposent des modifications chimiques sur l’ADN ou sur ses protéines associées ; voir section). C’est donc une fusion entre une Cas9 incapable de couper l’ADN, et une enzyme de modification de l’épigénome, qui sera recrutée sur la région génomique cible, et elle déposera les modifications chimiques voulues, sur la région génomique voulue (si on a fusionné à Cas9 une enzyme qui méthyle l’ADN, elle déposera, localement sur la région‑cible, des groupements méthyl sur son ADN ; de même si on a fusionné une enzyme qui acétyle les protéines associées à l’ADN, etc).
La simplicité d’utilisation de Cas9, sa grande efficacité, et sa capacité à réaliser des interventions qui étaient impossibles jusque-là (éditer le génome ou l’épigénome pratiquement à volonté), lui a valu un grand succès dans les laboratoires de recherche, qui avaient besoin de ce genre d’outils pour explorer les fonctions du génome et de l’épigénome. En quelques années, la technologie Cas9 a envahi pratiquement tous les laboratoires de biologie moléculaire du monde.
V. Un outil trop efficace ?
Cas9 est une protéine bactérienne, qui fonctionne sur l’ADN – or tous les êtres vivants portent leur information génétique sur de l’ADN. C’est ce qui explique le succès de la technologie Cas9 dans une grande variété d’espèces, pas uniquement chez les bactéries. Parmi les espèces où Cas9 permet de modifier l’information génétique, se trouve notamment l’être humain : le développement de cette technologie pourrait donc faciliter, très vraisemblablement, la modification génétique d’êtres humains.
La production d’organismes génétiquement modifiés est contrôlée, dans tous les États où elle est techniquement possible (c’est à dire, les États disposant d’infrastructures de recherche suffisamment développées). Notamment la production d’êtres humains génétiquement modifiés est interdite dans tous ces pays.
Dans certains pays toutefois, la recherche sur l’embryon humain est autorisée, du moment que les embryons modifiés ne sont ensuite pas implantés chez une mère porteuse (on ne leur permet pas de se développer pour donner un individu à naître). Il est donc possible, dans ces pays, d’essayer d’utiliser Cas9 pour modifier le génome humain, ce qui accélérerait la mise au point de la thérapie génique3. Des publications scientifiques commencent donc à décrire le résultat d’expériences où des embryons humains, porteurs de mutations provoquant des maladies génétiques, ont été traités par Cas9 pour corriger ces mutations.
À la fin de l’été 2017, une publication4 a ainsi présenté les résultats de ses essais de correction de mutation causant une maladie génétique, sur des embryons humains qui sont restés viables après traitement (et qui auraient donc pu être implantés dans une mère porteuse). Les statistiques étaient impressionnantes, elles suggéraient que la correction était très efficace. Les résultats méritent cependant d’être confirmés indépendamment : le raisonnement des auteurs de cette étude semble contenir quelques incohérences, et leur méthode de mesure de la correction de la mutation a certainement tendance à la surestimer5.
Même s’il s’avère que les auteurs ont effectivement surestimé le succès de leur méthode, il faut s’attendre à ce que, dans les prochaines années, de nouveaux articles, moins controversés, décrivent des modifications réussies du génome d’embryons humains. Il est donc utile de continuer à réfléchir aux implications de ces travaux sur la société : s’il devient possible de guérir des maladies génétiques avant implantation, est-il souhaitable de le faire ? Est-il préférable de modifier génétiquement un embryon porteur de la maladie, ou de sélectionner (parmi une collection d’embryons) les embryons qui ne sont pas porteurs, et n’implanter que ceux-là ? Plus généralement, quels sont les risques qu’apporte la possibilité d’éditer le génome humain, éventuellement hors des gènes connus pour provoquer des maladies génétiques ? La différence n’est d’ailleurs pas toujours claire entre la « correction d’un défaut » et l’« amélioration » (certaines interventions génétiques sembleraient difficiles à catégoriser ; par exemple, une modification génétique visant à modifier la couleur de la peau : il est établi que des peaux plus sombres protègent contre le cancer de la peau, mais la couleur de la peau a souvent été utilisée pour des discriminations dans les sociétés humaines).
Jusqu’à un passé récent, ces questions restaient essentiellement théoriques : il semblait tellement difficile de produire des êtres humains génétiquement modifiés, que les laboratoires susceptibles d’en être capables étaient rares, dépendaient beaucoup des financements de leurs gouvernements, et étaient tous situés dans des pays au gouvernement stablement démocratique. Mais la technologie Cas9 est tellement simple d’utilisation, qu’il devient possible que des laboratoires peu surveillés puissent mener ce genre de travaux : quel que soit le risque réel, il faut donc, au moins, considérer que ce risque est géographiquement étendu.
Outre de potentiels dangers, mal définis, dus à l’application de la technologie Cas9 au génome humain, il faut également s’interroger sur les conséquences irréversibles qu’elle pourrait avoir sur les écosystèmes.
L’édition du génome permet le développement de transgènes qui, au moins théoriquement, auraient la capacité d’envahir rapidement les populations naturelles, au point que l’introduction de quelques individus transgéniques dans le milieu naturel pourrait, après quelques cycles reproductifs, rendre transgénique la presque‑totalité de la population (voir Figure 5).
Fig. 5 : Un transgène invasif.
Chez les animaux, plantes et champignons, chaque individu possède deux versions de chaque chromosome, donc, de chaque gène (un chromosome est hérité de son père, l’autre de sa mère). On peut imaginer préparer, en laboratoire, des individus transgéniques, qui portent deux copies du transgène (ici en rouge), et où le transgène est constitué du gène de la Cas9 et du gène d’un ARN-guide dirigé contre la version naturelle du gène situé dans cette région chromosomique (la version naturelle est représentée en gris). Si on lâche ces individus transgéniques dans la nature, qu’ils se croisent avec des individus sauvages de sexe opposé, leur descendance portera les deux versions du chromosome : une version transgénique, qui permet d’exprimer la Cas9 et l’ARN-guide, et une version sauvage. Dans les cellules de ce descendant, il sera donc possible que Cas9, guidée par l’ARN, coupe la version naturelle du gène. Dans certaines cellules, la coupure n’aura pas lieu, ou alors, elle aura lieu mais sera réparée sans copier le transgène (flèche de gauche) : dans ces cellules, la proportion entre transgène et gène naturel ne sera pas modifiée. Dans certaines cellules en revanche (flèche de droite), le gène naturel sera coupé sous l’effet de Cas9, et il sera réparé en utilisant le transgène comme modèle (puisque le transgène se trouve dans la même région génomique, il est flanqué des mêmes séquences d’ADN de part et d’autre, donc le mécanisme de réparation pourra détecter le transgène comme « similaire » à la séquence coupée, et l’utiliser comme modèle pour la réparation). À la fin du processus, ces cellules porteront donc deux copies du transgène, contre une seule initialement. Si ce phénomène se produit dans des cellules germinales, 100 % des gamètes seront transgéniques (on appelle « gamètes » les cellules germinales matures : chez les animaux, ce sont les spermatozoïdes chez les mâles, les ovules chez les femelles). Finalement, le transgène a la propriété de remplacer, dans le génome, la copie naturelle du gène, et donc de se transmettre plus efficacement que lui à la descendance des générations suivantes (l’ampleur de cet avantage dépend de la fréquence la coupure et de la réparation par copie du transgène, dans les cellules germinales).
En théorie au moins, Cas9 permet donc de produire des transgènes qui devraient se multiplier dans les populations (alors qu’un transgène normal, s’il ne donne pas d’avantage ou d’inconvénient particulier par rapport aux individus sauvages, reste en proportion stable au cours des générations). Si bien qu’après l’introduction, dans l’écosystème, d’un nombre limité d’individus transgéniques, la presque‑totalité de la population sauvage sera devenue transgénique au bout de plusieurs générations.
Ce genre de perspective a de quoi effrayer, on se demande pourquoi des gens réfléchissent à la préparation de transgènes qui auraient, de cette manière, le pouvoir de modifier de façon irréversible un écosystème. Les introductions d’espèces par l’homme se sont souvent, par le passé, traduites en catastrophes pour les espèces locales : alors si la modification est volontairement rendue plus efficace et irréversible, on peut s’attendre à des conséquences encore pires...
Pourtant des laboratoires envisagent sérieusement d’utiliser ce type de transgène pour modifier les populations naturelles6 pour un tour d’horizon, en anglais, des caractéristiques de ces projets en 2014). C’est que certaines applications semblent tentantes, notamment en ce qui concerne la lutte contre le paludisme (ou : malaria). Pour les humains, c’est la maladie qui tue le plus, elle est due à un parasite qui est transmis par une piqûre de moustique. Seules certaines espèces de moustiques peuvent le transmettre (les autres espèces détruisent le parasite quand elles ingèrent le sang d’une personne infectée). On connaît maintenant plusieurs gènes qui affectent la faculté de transmission du parasite par le moustique. Si on pouvait remplacer, dans la population naturelle de moustiques, l’un de ces gènes par autre chose (ici : par un transgène exprimant Cas9 et un ARN‑guide dirigé contre ce gène), on supprimerait la transmission de la maladie – au prix d’une modification limitée de l’écosystème : on n’a pas supprimé les moustiques, on ne les a même pas remplacés par une autre espèce de moustiques, on les a simplement remplacés par des moustiques de la même espèce, mais où ce gène a été inactivé.
De tels transgènes, conçus pour remplacer un gène donné dans une population naturelle, offrent donc la possibilité de sauver des centaines de milliers de vies humaines par année, sans faire peser sur les écosystèmes le poids des méthodes classiques (épandages d’insecticides, introduction de prédateurs). Finalement, l’impact écologique serait probablement moindre que celui des méthodes actuelles.
La préparation de telles souches de moustiques transgéniques est simple, elle a même, certainement, déjà été réalisée en laboratoire, et il suffit d’une décision politique pour que ces souches soient introduites dans le milieu naturel. Il s’agit donc de faire un choix : doit‑on prendre le risque de modifier, de façon probablement irréversible, les populations naturelles de moustiques ? Ou doit‑on s’abstenir, au prix possible d’un million de morts humaines par an ? Pour l’heure, ces introductions ne sont pas autorisées. Les laboratoires de recherche, et leurs décideurs, se trouvent dans des pays occidentaux, qui ne sont pas touchés par la maladie, et il est probable que les populations concernées aient une opinion différente. D’autant que, confrontés au même problème il y a quelques décennies, les pays occidentaux ne se sont pas embarrassés des mêmes scrupules : la Corse était touchée par le paludisme jusqu’au milieu du xxe siècle, et c’est par des épandages massifs de DDT qu’elle en a été débarrassée7. Cet insecticide chimique, à longue persistance dans l’environnement, et toxique pour de nombreuses espèces non‑ciblées, n’a pas suscité à l’époque les mêmes hésitations que l’introduction de ces moustiques transgéniques aujourd’hui.
La question est donc, évidemment, compliquée (elle aurait déjà été résolue si elle avait été simple), et la décision est rendue d’autant plus difficile qu’il ne semble pas possible de faire une expérience pour mesurer les conséquences d’une telle introduction de transgène invasif : la seule expérience raisonnablement convaincante serait son introduction effective dans la nature.
Conclusion
Cas9 est une enzyme qui peut couper l’ADN de façon spécifique : guidée par un ARN de séquence arbitraire, elle va couper les régions d’ADN qui présentent un segment d’identité suffisamment long à son ARN‑guide. Elle constitue donc une enzyme programmable, qui peut cibler, avec très peu de contraintes, des régions génomiques d’intérêt.
En recherche fondamentale, elle a été adoptée en l’espace de quelques années, et fait maintenant partie des outils employés en routine pour l’analyse des fonctions du génome et de l’épigénome.
Sa mise en œuvre pratique, qui s’appuie sur des méthodes développées précédemment pour d’autres types d’expériences, est assez simple – au point qu’elle suscite des craintes inédites : il est devenu concevable que des laboratoires peu contrôlés, ou que des États peu démocratiques, la mettent à profit pour des applications dangereuses.
Devant cet état de fait, la communauté scientifique doit s’assurer que ses travaux ne serviront pas des intérêts contraires à l’intérêt général. Même s’il est loin d’être établi que la technologie CRISPR/Cas9 puisse avoir des applications dangereuses, il est nécessaire d’envisager la possibilité qu’elle en ait.
Faut-il, alors, arrêter la recherche sur ce sujet ? D’une certaine manière, il est déjà trop tard : comme toujours, la science a progressé de manière incrémentale, et les applications actuelles de cette technologie étaient inconnues des chercheurs qui ont identifié les régions CRISPR dans les génomes bactériens à la fin du xxe siècle, ou qui ont analysé leur mode d’action au début du xxie siècle. Les spéculations sur les dangers possibles de cet outil ont commencé alors que les données scientifiques étaient déjà disponibles depuis de nombreuses années, et accessibles à d’éventuels utilisateurs mal intentionnés.
Dans ces conditions, la meilleure conduite à tenir est probablement de s’assurer que les laboratoires publics, contrôlés par des gouvernements raisonnables, gardent le leadership de la maîtrise de cette technologie, au cas où elle serait vraiment dangereuse. Les meilleurs experts du sujet sont, actuellement, employés par des institutions de recherche publiques : il est important qu’ils le restent, ne serait‑ce que pour que les pouvoirs publics gardent la capacité de comprendre ce qui se fait ailleurs (dans des groupes privés ou, sur un autre plan, dans des organisations hostiles). Même si cette technologie peut avoir des applications néfastes, et peut‑être : parce que cette technologie pourrait avoir des applications néfastes, il est nécessaire que la recherche publique continue à s’y intéresser.